TL;DR
Гайд по тому, как собрать базу научных статей, скачать им всем полный текст в формате PDF и загрузить в LLM с большим контекстным окном. По сути, это контролируемый аналог Academic Pro Search от Perplexity. Через базу данных OpenAlex скачиваем интересующие нас статьи в формате .ris, конвертируем их в .json и загружаем в Google AI Studio. Там устанавливаем температуру на ноль, выбираем Gemini 2.5 Pro и просим выбрать 50 наиболее релевантных статей. Скачиваем список в формате .bib, загружаем в Zotero и ищем полные тексты в формате PDF. Найденные PDF загружаем в NotebookLM, где задаем вопросы.
Введение
Всем привет! Меня зовут Толя, и я профессиональный учёный. Да-да, по смешному стечению обстоятельств, я единственный "Scientist" в своём НИИ на 500 человек, остальные у нас имеют должность "Researcher". Разницы особой нет, мы все так же пишем статьи и занимаемся "Науками о Земле". А писать статьи я люблю, правда, пока получается непродуктивно, за 8 лет опубликовал 38 статей, которые процитировали всего 300 раз. Так что в научном мире я примерно как ФК Тоттенхэм: "вроде и говном ты не был, но и до высот не добрался".
Представьте, что вам для работы, статьи или просто праздного любопытства нужно найти ответ на вопрос, и чтобы этот ответ был подкреплён научными исследованиями. Если это ваша область интересов, то, скорее всего, вы уже знаете источники, где искать ответ. А что если это смежная область или совсем что-то новое? Что если в момент написания заявки на грант или научной статьи про собак вы задались вопросом: "от чего зависит количество рождающихся котят у кошек?".

В таких случаях долгое время для меня готовым решением было использовать Perplexity или Consensus (коротко о них писал тут). Потом ChatGPT научился поиску по интернету, и это стало неплохим подспорьем. Все три варианта быстро справляются с задачей, однако обладают большим минусом, который меня очень сильно раздражал за годы использования. Они ищут статьи в базах данных PubMed и Semantic Scholar, что само по себе ОК, но почему-то в области "Науки о Земле" там доминируют статьи из журналов издательств MDPI и Frontiers (почитайте тут, почему это плохо). Такие ссылки не только стыдно добавить в список литературы, но их ещё и мало, обычно 10-15 максимум. Не говоря уже про ошибки, свойственные LLM (так называемые "галлюцинации").
В какой-то момент я задался вопросом: а как, собственно, сделать поиск по проверенным статьям? Я думаю, что многие слышали о NotebookLM, и это, по сути, является ответом. Но возникает следующий вопрос: а как найти статьи, по которым делать поиск? Одно дело, когда у вас есть подборка любимых статей и книг, или СНиПов. А что если такого нет? В данной заметке я как раз предлагаю рассмотреть одно такое решение.
Нам понадобятся
- Zotero 7 – менеджер библиографических ссылок, в данном гайде нужен только для того, чтобы скачивать PDF статей. Я рекомендую поставить Zotero 7 Beta, так как на стабильной версии постоянно возникает капча при скачивании статей. Если знаете другой способ сделать это быстро и не сталкиваясь с капчей, то дайте, пожалуйста, знать.
- Zotero Attanger – расширение Zotero, которое позволяет хранить файлы не в Zotero (у него есть ограничение в 300 Мб), а в любой директории на диске. Настраивается по этому гайду. Если коротко, то бесплатные 300 Мб будут использоваться для синхронизации текстовых файлов и метаданных, сами PDF будут храниться в виде ссылок.
- Аккаунт Google, и доступ к NotebookLM и Google AI Studio. Бесплатный аккаунт подойдет, единственное, что в нем есть ограничения на 50 документов для NotebookLM, ПРО-подписка увеличит лимит до 300 документов.
Я буду описывать no-code решение, однако, всё, конечно, можно закодить. Так что для любителей повозиться с автоматизацией я в конце предлагаю частичную реализацию.
Поиск литературы
В первую очередь, надо найти литературу, с которой мы будем в дальнейшем работать. Я буду рассматривать ситуацию, где пользователь слабо знаком с областью, просто имеет общие представления о том, что такое хорошая статья, а что лучше не открывать. На данном этапе наша задача собрать максимум литературы, которая была опубликована по интересующей нас теме.
Для этого идем в любой поисковик по базам данных цитирований. Подойдет Scopus, Web of Science, Google Scholar, PubMed или Semantic Scholar. В качестве примера буду использовать OpenAlex (OA), так как он бесплатный, быстрый и с очень большим лимитом на экспорт. Кроме того, доступен API для продвинутых. На сайте выполняем поиск по топику, для меня это "quantile mapping". Далее фильтруем результат, я ограничил годом 2010, чтобы это была именно статья в журнале (а не книга), чтобы ее хотя бы раз процитировали, язык английский и т. д. И самое главное, выбираем издателей, которым доверяете. Я обычно предпочитаю издателей из большой пятерки Elsevier, Taylor & Francis, Wiley, Springer и SAGE, плюс Copernicus. В зависимости от вашего уровня этичности, либо ставим Open-Access Only, либо не ставим.
Фильтр по издательствам может быть очень суровым и не убережет вас от низкокачественных публикаций. В истории много прецедентов, где уважаемые десятилетиями журналы ловились на недобросовестных публикациях и за это были наказаны. Самый громкий за последние годы это Chemosphere.
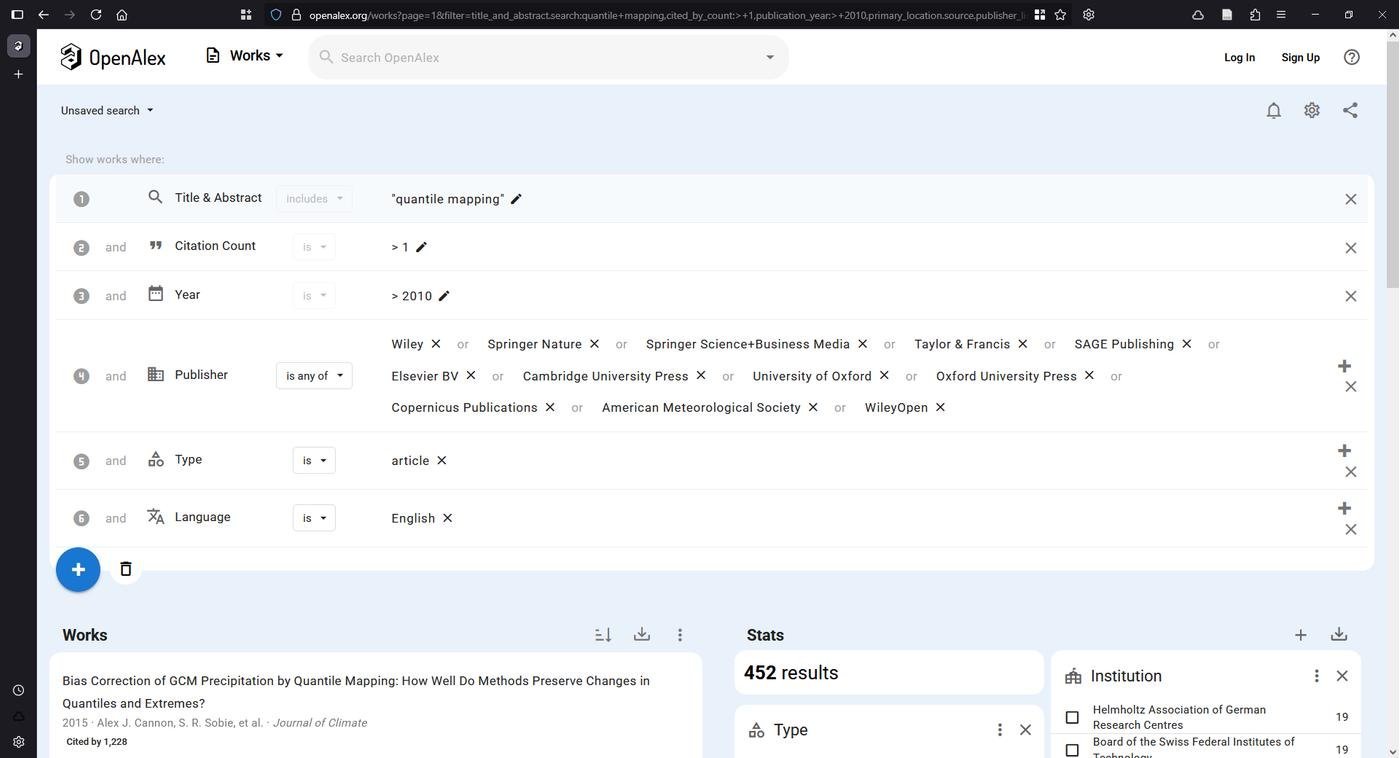
В моем случае выборка составила 452 статьи. Важно отметить, что это просто статьи, где либо в названии, либо в абстракте, содержалась фраза "quantile mapping". Я специально не ограничивал выборку по топикам OA, так как мне кажется, что это может оказать медвежью услугу и удалить лишнее, поскольку неизвестно, кто эти топики назначал. Вместо этого я предлагаю воспользоваться LLM с думающим режимом (reasoning-модели короче). Для этого скачиваем нашу выборку из OA в формате .ris и любым удобным способом перегоняем в .json. Я импортировал этот .ris в отдельную коллекцию в Zotero, а затем сделал экспорт в Better CSL Json.
Фильтрация выборки
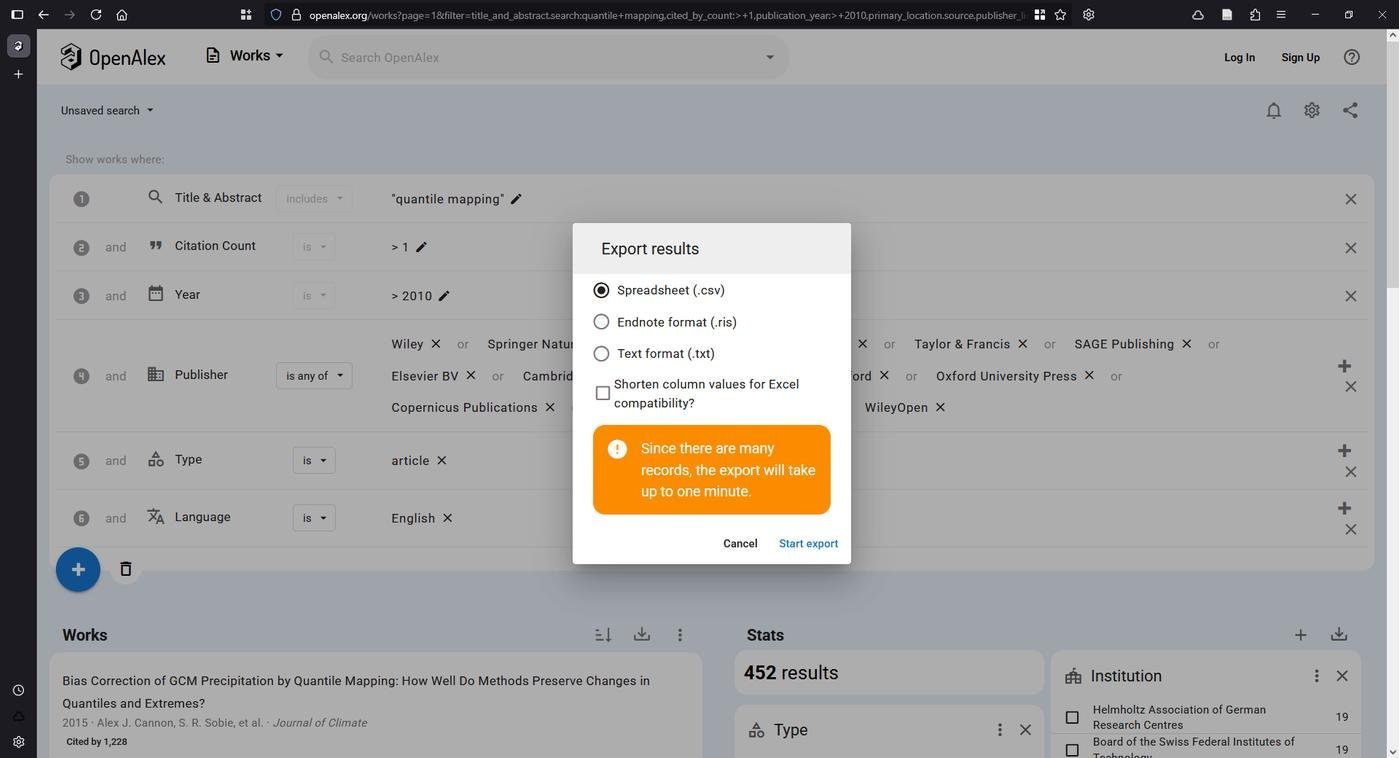
С полученным .json идем в Google AI Studio. Выбираем Gemini 2.5 Pro, ставим температуру на 0 и загружаем файл. Такие настройки ограничат LLM только своим контекстным окном и снизят вероятность ошибки. Моя выборка из 452 статей заняла всего 32% контекстного окна Gemini (322,318 / 1,048,576 токенов). Теперь пишем простенький запрос-просьбу отфильтровать только наиболее подходящие по теме и выдать их в формате BibTeX, чтобы было удобно работать со списком дальше. К сожалению, вывод ограничен 65,536 токенами, так что очень легко упереться в лимиты. Но 50 статей в формате BibTeX без абстрактов влезают. У меня правда, получилось почему-то 94 статьи, а не 50, но да ладно, в токены на вывод я влез.
Полученный список копируем в любой текстовый редактор (Блокнот, Vim, Sublime и т.д.) и сохраняем в файл расширения .bib, в моем случае это qmap.bib
Поиск полного текста статей
Сначала надо запустить Zotero и создать там коллекцию (или папку, называйте как хотите), куда статьи будут скачиваться. Например, "qmap" в корне. Переходим в нее и импортируем файл qmap.bib через меню File → Import. Убираем галочку с "Place imported collections and items into new collection", так как иначе у нас создастся подпапка в нашей папке.
Выделяем все статьи в нашей подборке (Ctrl/Cmd + A работает). Кликаем правой кнопкой мыши на выделение и нажимаем "Find Full Text". Всё, Zotero начал искать PDF в открытом доступе, скачивать их, а Zotero Attanger, в свою очередь, перемещать их куда нужно. В зависимости от вашего соединения, доступа к библиотеке, этот процесс может занять какое-то время. Какие-то могут не найтись, что ж, "Nobody is perfect..."
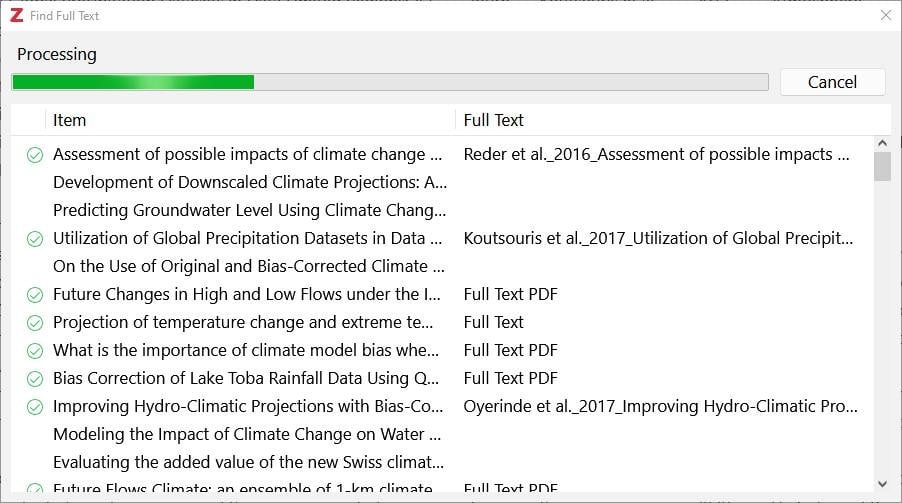
"... my name is Nobody". Чтобы найти как можно больше PDF, можете поставить расширение scipdf, которое неэтично расширяет возможности поиска. У меня получилось таким образом получить 82/94 статей. Для предварительного анализа самое то.
Подитог
Таким образом, мы получили какой-то state-of-the-art исследований, опубликованных в уважаемых журналах, цитируемых и отвечающие нашему топику. Для большинства статей получилось найти полный текст.
Анализ
Теперь задача загрузить и проанализировать эти документы в NotebookLM от Google. Начиная с мая 2025 года он работает на Gemini 2.5 Flash и поддерживает до 50 (300 в PRO-версии) документов. Также неплохо самостоятельно дополняет список источников своими PDF-ками, которые, я полагаю, берёт из тех доков, которые раньше загружали другие пользователи.
Если документов получилось не очень много, возможно, лучше пойти в Google AI Studio и попробовать пообщаться с Gemini 2.5 Pro, выставив температуру на ноль. Но это гораздо медленнее и нестабильнее. У меня так и не получилось нормально обработать все источники, постоянно ошибка то лимитов, то какая-то неизвестная.
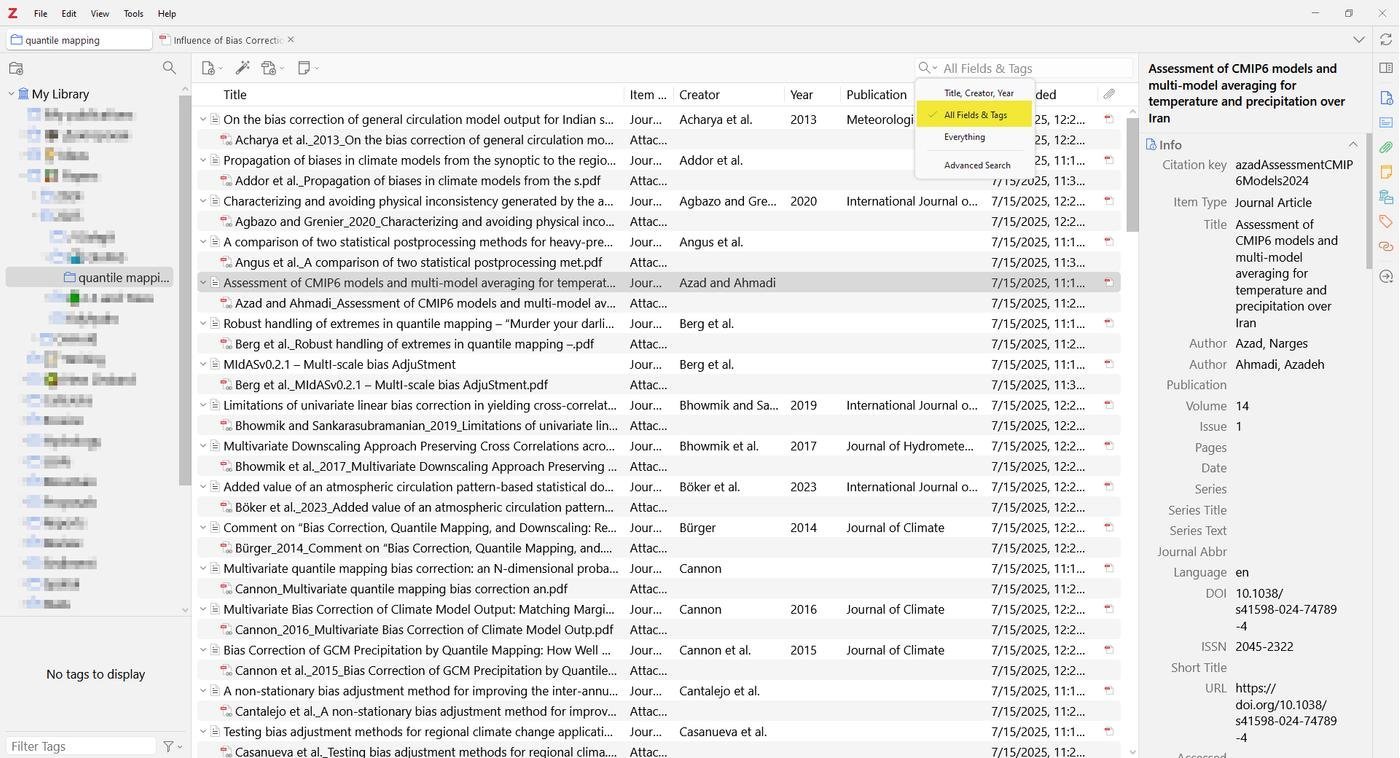
Чтобы загрузить PDF из Zotero в NotebookLM, надо сначала их скопировать в отдельную папку, так как по умолчанию файлы разбросаны по куче подпапок. Для этого идём в Zotero в нашу коллекцию и делаем поиск по "PDF" в окне поиска по "All Fields & Tags". Такая процедура покажет все файлы, которые есть в коллекции. Их надо выделить (Ctrl/Cmd + A) и перетащить в какую-нибудь папку в проводнике (Explorer/Finder), например во временную папку на рабочем столе. Это скопирует файлы из директории, где хранит их Zotero. Теперь все файлы в этой папке перетаскиваем в NotebookLM. Ждём загрузку и индексацию.
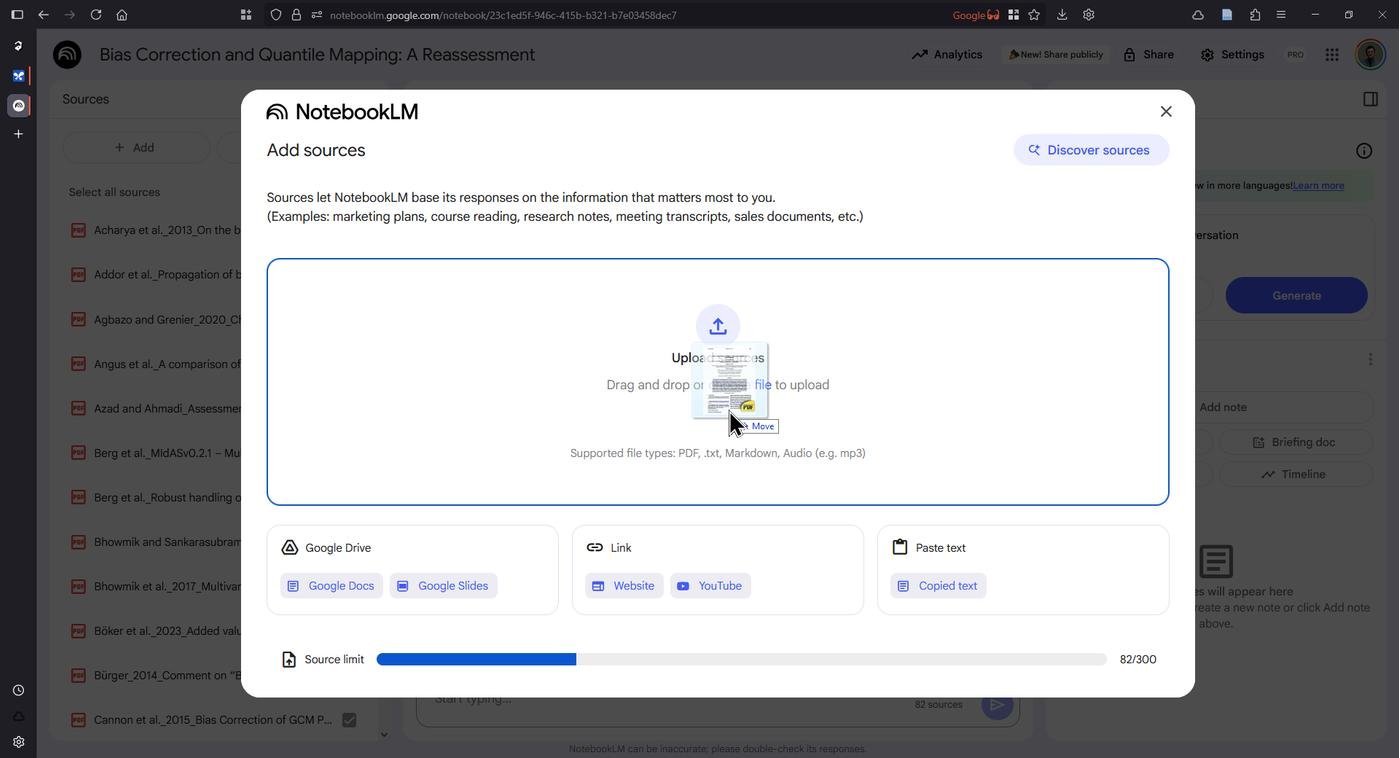
Как и с любой LLM, возможно, стоит прописать системный промпт, чтобы получить улучшенный результат. В настройках чата можно настроить "Conversation style", но там ограничения всего в 500 символов. Я обычно пользуюсь "System Prompt Generator for Reasoning Models", кастомным ChatGPT, который создал Денис Sexy IT. По умолчанию он выдаёт очень длинный системный промпт, на данном этапе важно попросить ограничиться 500 символами.
И это, по сути, всё. У вас на руках одна из самых мощных моделей (на июль 2025 г.) с щедрым бесплатным тиром и контекстом, настроенным специально под вашу задачу. Дальше всё зависит от вашего вопроса. Каждый ответ будет подтверждён ссылками на конкретный документ с точностью до абзаца. А благодаря тому, что использовался Zotero Attanger с умным переименованием, все документы имеют говорящее название.
NotebookLM умеет и на других языках общаться, это настраивается через Settings → Output Language
Таким же образом можно найти и выкачать все статьи, которые процитировали другую статью, и посмотреть, как изменялся дериватив. Или посмотреть, как используют вашу питоновскую библиотеку в научных статьях. Или проанализировать публикационную активность какого-то конкретного автора.
Убираем за собой
Если вы, так же как и я, с трепетом относитесь к своей библиотеке Zotero, то рекомендую после всех операций удалить ненужные статьи. Сначала статьи, которые показались интересными, перетаскиваем в другую коллекцию из нашей временной коллекции "qmap". Потом всю коллекцию "qmap" экспортируем в формат Better BibTeX в qmap.bib (ну или в .json экспортировать). Этот шаг нужен для того, чтобы идентифицировать пути, где сохранены неинтересные нам PDF. Далее на любом скриптовом языке пишем цикл, который пройдётся по всем PDF-файлам и удалит их, а затем удалит пустые папки. Например, на R можно так:
library(bib2df) # ropensci.r-universe.dev/bib2df
# Читаем BibTeX как датафрейм
biblib <- bib2df::bib2df("qmap.bib") |>
subset(!is.na(FILE))
# Удаляем все ненужные PDF
all(fs::file_exists(biblib$FILE))
fs::file_delete(biblib$FILE)
# Удаляем пустые папки
# Абсолютный путь до библиотеки Zotero
# (тот, который настроили при установки Zotero Attanger)
all_dirs <-
fs::dir_ls(
"C:\\Users\\TsyplenkovA\\OneDrive - MWLR\\ATS\\Personal\\zotero-library"
)
# Помечаем какие папки пустые
empty_dirs <-
vapply(
all_dirs,
\(x) {
length(fs::dir_ls(x)) == 0
},
FUN.VALUE = logical(1L)
)
# Удаляем !
names(empty_dirs[empty_dirs]) |>
unlink(force = TRUE, recursive = TRUE)
Created on 2025-07-15 with reprex v2.1.1
Автоматизация
Этап с поиском литературы можно автоматизировать, например, написав API запрос через библиотеки на R, Python, Julia или Kotlin. Но интереснее всего, что если скачать с сайта OA выборку в формате CSV, то там будет много рабочих URL на статьи, и можно пропустить шаг с Zotero.
Вы наверное думаете, во дурак, использует R. Так вот, у меня есть целый телеграм канал "Типизированный R" посвященный этому языку программирования. Подписывайтесь, там я очень медленно и пытаюсь показать, что R это настоящий язык программирования.
library(data.table)
library(dplyr)
library(curl)
# Читаем скачанную базу данных
df <- fread("works-2025-07-14T22-10-24.csv") |> as_tibble()
# Находим URL статей
urls <- df |>
select(pdf_url = best_oa_location.pdf_url) |>
filter(pdf_url != "") |>
pull(1)
# Скачиваем в корень рабочий директории
for (i in urls) {
tmp <- tempfile(tmpdir = ".", fileext = ".pdf")
tryCatch(
{
curl_download(i, tmp)
print(tmp)
},
error = function(e) FALSE
)
}
#> [1] ".\\file44a0fbd4d7.pdf"
#> [1] ".\\file44a01478278.pdf"
Created on 2025-07-15 with reprex v2.1.1
Почему "неэтичный"?
Я назвал такой подход "неэтичным" по двум причинам. Во-первых, из простого поиска ответа на вопрос легко можно сделать полноценный литобзор. Такое публиковать пожалуй не стоит, язык LLM легко распознаётся, посмотрите последнюю статью Дмитрия Кобака на этот счёт. Во-вторых, получение полных текстов статей при помощи scipdf всё-таки нарушение авторских прав и крайний случай. И делать так не стоит, если вы, конечно, не "новый русский пират".





Классно, очень интересно!
Весьма толково и подробно описано, благодаря чему воспроизводится за считанные минуты, в моем кейсе Zotero правда всего 2 статьи из 15 скачать смог, без неэтичных плагинов, но это уже нюансы. Очевидно более правильно будет урлы из CSV вытаскивать, как было упомянуто.
Строили у себя похожий пайплайн для анализа патентов, очень близкая тема и многие процессы прям одинаковые вплоть до последней запятой.
Эта задача отлично "мап-редьюсится": RIS файл аккуратно нарезается на мелкие кусочки, каждый из которых скармливается LLM с одним и тем же запросом, результаты уже потом склеиваются в BIB (или не склеиваются и импортируются отдельно).
Таким образом решается проблема длины контекста, так что можно адаптировать длину чанка под любые доступные модели. А также убирается проблема поиска иголки в стоге сена. Все-таки длинный контекст в лям токенов труднее эффективно обработать, даже для Gemini, чем более короткий и конкретный фрагмент.
Примерно тоже самое можно использовать, если по теме нашлось более трехсот публикаций и NotebookLM сказал "Ой!"
Ээээ... если это - "непродуктивно", то что же тогда "продуктивным" считается? :О
Автор, большое спасибо!
Прочитал, узнал много нового, положил в закладки, что-то возьму на вооружение.
Ничего не понятно, но очень интересно 😁Плюсик в карму
Большое спасибо! Попробовал сделать обзор по гайду, для поверхностного ознакомления с темой нормас. Но заметил, что в NotebookLM в итоговом тексте нейронка ссылается по сути на штук 5 статей и часто в качестве источника она показывает просто название статьи в списке литры. Так что промт нужно очень тщательно продумывать и, возможно, грузить статьи меньшими "батчами".